Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
廃炉環境国際共同研究センター; 慶應義塾*
JAEA-Review 2021-048, 181 Pages, 2022/01
日本原子力研究開発機構(JAEA)廃炉環境国際共同研究センター(CLADS)では、令和2年度英知を結集した原子力科学技術・人材育成推進事業(以下、「本事業」という)を実施している。本事業は、東京電力ホールディングス株式会社福島第一原子力発電所(1F)の廃炉等をはじめとした原子力分野の課題解決に貢献するため、国内外の英知を結集し、様々な分野の知見や経験を、従前の機関や分野の壁を越えて緊密に融合・連携させた基礎的・基盤的研究及び人材育成を推進することを目的としている。平成30年度の新規採択課題から実施主体を文部科学省からJAEAに移行することで、JAEAとアカデミアとの連携を強化し、廃炉に資する中長期的な研究開発・人材育成をより安定的かつ継続的に実施する体制を構築した。本研究は、研究課題のうち、令和元年度に採択された「微生物生態系による原子炉内物体の腐食・変質に関する評価研究」の令和元年度と令和2年度の研究成果について取りまとめたものである。本課題は令和2年度が最終年度となるため2年度分の成果を取りまとめた。本研究の目的は、福島第一原子力発電所の廃炉プロセスに有用となる微生物に関係した知見を得ることにある。このため、同発電所やその敷地内外に生息する微生物群集の実態を明らかにする。1Fの敷地境界南(処理水タンク群の南)の表層土、発電所近くの海底土とその直上水、3km沖合の表層水等からサンプルを採取し、メタゲノム解析(微生物の培養を介せず、そのDNAを直接解読することで、生息する微生物の情報を得ること)を実施した。その結果、現状で、1F敷地周辺で検出される放射線量であれば、その高低にかかわらず、同じような環境を比較した場合、細菌叢の構造に大きな変化がないことが示唆された。また、1F2号機のトーラス室に由来する環境DNAの解析を行い、トーラス室では、チオ硫酸塩酸化細菌が主たる構成細菌として同定されると共に、幾つかの細菌種がバイオフィルム(微生物の集合体)を作っている可能性を示唆した。共同研究を行ったロシアのカザン大学の研究者は、日本で得られた配列データを情報学的に解析すると共に、ロシアの放射線による環境汚染に関してまとめた。これらの知見を総括し、1Fの廃炉プロセスに有用となる提言をまとめた。
廃炉環境国際共同研究センター; 慶應義塾大学*
JAEA-Review 2020-047, 63 Pages, 2021/01
日本原子力研究開発機構(JAEA)廃炉環境国際共同研究センター(CLADS)では、令和元年度英知を結集した原子力科学技術・人材育成推進事業(以下、「本事業」という)を実施している。本事業は、東京電力ホールディングス福島第一原子力発電所の廃炉等をはじめとした原子力分野の課題解決に貢献するため、国内外の英知を結集し、様々な分野の知見や経験を、従前の機関や分野の壁を越えて緊密に融合・連携させた基礎的・基盤的研究及び人材育成を推進することを目的としている。平成30年度の新規採択課題から実施主体を文部科学省からJAEAに移行することで、JAEAとアカデミアとの連携を強化し、廃炉に資する中長期的な研究開発・人材育成をより安定的かつ継続的に実施する体制を構築した。本研究は、研究課題のうち、「微生物生態系による原子炉内物体の腐食・変質に関する評価研究」の令和元年度の研究成果について取りまとめたものである。本研究の目的は、福島第一原子力発電所の廃炉プロセスに有用となる微生物に関係した知見を得ることにある。このため、同発電所やその敷地内外に生息する微生物群集の実態を明らかにする。令和元年度は、敷地境界南(処理水タンク群の南)の表層土、発電所近くの海底土とその直上水、3km沖合の表層水等からサンプルを採取し、各環境DNAの取得に成功した。環境DNAの塩基配列を決定することで、主にバクテリアと微細藻類における生物群集を明らかにした。また、ロシアのカザン大学との共同研究を開始した。
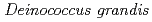 , isolated from freshwater fish in Japan
, isolated from freshwater fish in Japan佐藤 勝也; 小野寺 威文*; 面曽 宏太*; 武田-矢野 喜代子*; 片山 豪*; 大野 豊; 鳴海 一成*
Genome Announcements (Internet), 4(1), p.e01631-15_1 - e01631-15_2, 2016/01
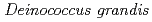 was isolated as a gram-negative, red-pigmented, radioresistant, rod-shaped bacterium from freshwater fish. The draft genome sequence of
was isolated as a gram-negative, red-pigmented, radioresistant, rod-shaped bacterium from freshwater fish. The draft genome sequence of 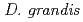 ATCC 43672 was 4,092,497 bp, with an average G+C content of 67.5% and comprised of 4 circular contigs and 3 linear contigs that was deposited at DDBJ/EMBL/Genbank under the accession number BCMS00000000. The draft genome sequence indicates that
ATCC 43672 was 4,092,497 bp, with an average G+C content of 67.5% and comprised of 4 circular contigs and 3 linear contigs that was deposited at DDBJ/EMBL/Genbank under the accession number BCMS00000000. The draft genome sequence indicates that 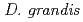 possesses a DNA damage response regulator (encoded by
possesses a DNA damage response regulator (encoded by 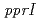 homolog) and radiation-desiccation response regulons (
homolog) and radiation-desiccation response regulons (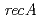 ,
, 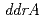 ,
, 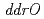 ,
, 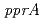 and
and 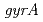 homologs etc.), which are involved in the unique radiation-desiccation response system in
homologs etc.), which are involved in the unique radiation-desiccation response system in 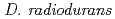 . As it is for
. As it is for 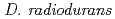 ,
, 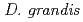 seems to employ the same radioresistant mechanisms. In future, the draft genome sequence of
seems to employ the same radioresistant mechanisms. In future, the draft genome sequence of 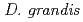 will be useful for elucidating the common principles of the radioresistance based on the extremely efficient DNA repair mechanisms in Deinococcus species by the comparative analysis of genomic sequences.
will be useful for elucidating the common principles of the radioresistance based on the extremely efficient DNA repair mechanisms in Deinococcus species by the comparative analysis of genomic sequences.
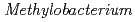 sp. ME121, isolated from soil as a mixed single colony with
sp. ME121, isolated from soil as a mixed single colony with 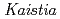 sp. 32K
sp. 32K藤浪 俊*; 武田 喜代子*; 小野寺 威文*; 佐藤 勝也; 清水 哲*; 若林 佑*; 鳴海 一成*; 中村 顕*; 伊藤 政博*
Genome Announcements (Internet), 3(5), p.e01005-15_1 - e01005-15_2, 2015/09
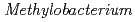 sp. ME121 was isolated from soil as a mixed single colony with
sp. ME121 was isolated from soil as a mixed single colony with 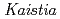 sp. 32K during our screening of L-glucose-utilizing microorganisms, and its growth was enhanced by coculture. It was expected that genomic analysis of this bacterium would provide novel information on coculture-dependent growth enhancement. The genomic information of symbiotic bacteria could be of use for studying the molecular mechanisms underlying microbial symbiosis. The draft genome sequence of
sp. 32K during our screening of L-glucose-utilizing microorganisms, and its growth was enhanced by coculture. It was expected that genomic analysis of this bacterium would provide novel information on coculture-dependent growth enhancement. The genomic information of symbiotic bacteria could be of use for studying the molecular mechanisms underlying microbial symbiosis. The draft genome sequence of 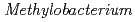 sp. ME121 is 7,096,979 bp in total length and comprises 197 large contigs (
sp. ME121 is 7,096,979 bp in total length and comprises 197 large contigs ( 500 bp) that was deposited at DDBJ/EMBL/GenBank under the accession number BBUX00000000. The draft genome sequence shows that
500 bp) that was deposited at DDBJ/EMBL/GenBank under the accession number BBUX00000000. The draft genome sequence shows that 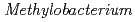 sp. ME121 has some genes that encode putative methanol/ethanol family PQQ-dependent dehydrogenases involved in methylotrophy. Some unknown factor provided by the coculture may contribute to increase the growth of
sp. ME121 has some genes that encode putative methanol/ethanol family PQQ-dependent dehydrogenases involved in methylotrophy. Some unknown factor provided by the coculture may contribute to increase the growth of 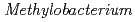 sp. ME121.
sp. ME121.
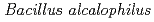 AV1934, a classic alkaliphile isolated from human feces in 1934
AV1934, a classic alkaliphile isolated from human feces in 1934Attie, O.*; Jayaprakash, A.*; Shah, H.*; Paulsen, I. T.*; 守野 正人*; 高橋 優嘉*; 鳴海 一成*; Sachidanandam, R.*; 佐藤 勝也; 伊藤 政博*; et al.
Genome Announcements (Internet), 2(6), p.e01175-14_1 - e01175-14_2, 2014/11
The alkaliphilic bacterium 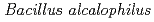 AV1934, isolated from human feces, recent has indicated novel potassium ion coupling to motility in this extremophile. We report draft sequences that will facilitate an examination of whether that coupling is part of a larger cycle of potassium ion-coupled transporters. This draft genome has 4,095 predicted genes and 4,063 predicted proteins. The whole-genome shotgun project has been deposited in Genbank under the accession no. ALPT02000000. In addition to the
AV1934, isolated from human feces, recent has indicated novel potassium ion coupling to motility in this extremophile. We report draft sequences that will facilitate an examination of whether that coupling is part of a larger cycle of potassium ion-coupled transporters. This draft genome has 4,095 predicted genes and 4,063 predicted proteins. The whole-genome shotgun project has been deposited in Genbank under the accession no. ALPT02000000. In addition to the 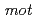 genes encoding the MotPS channel that uses either potassium or sodium coupling, two loci encoding multisubunit Mrp-type antiporters, were found. The
genes encoding the MotPS channel that uses either potassium or sodium coupling, two loci encoding multisubunit Mrp-type antiporters, were found. The 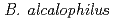 AV1934 genome reveals no tripartite ATP-independent (TRAP-T family) uptake systems, a major sodium-coupled complement in the other two alkaliphiles. The
AV1934 genome reveals no tripartite ATP-independent (TRAP-T family) uptake systems, a major sodium-coupled complement in the other two alkaliphiles. The 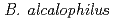 AV1934 genomic data will enable us to test the hypothesis of a major role of potassium in ion coupling in this extremophile.
AV1934 genomic data will enable us to test the hypothesis of a major role of potassium in ion coupling in this extremophile.
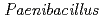 sp. strain TCA20, isolated from a hot spring containing a high concentration of calcium ions
sp. strain TCA20, isolated from a hot spring containing a high concentration of calcium ions藤浪 俊*; 武田 喜代子*; 小野寺 威文*; 佐藤 勝也; 佐野 元彦*; 高橋 優嘉*; 鳴海 一成*; 伊藤 政博*
Genome Announcements (Internet), 2(5), p.e00866-14_1 - e00866-14_2, 2014/09
Calcium-dependent 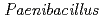 sp. strain TCA20 was isolated from a water sample of a hot spring containing a high concentration of calcium ions. Here, we report the draft genome sequence of this bacterium, which may be the basis for the research of calcium ion homeostasis. The draft genome sequence of
sp. strain TCA20 was isolated from a water sample of a hot spring containing a high concentration of calcium ions. Here, we report the draft genome sequence of this bacterium, which may be the basis for the research of calcium ion homeostasis. The draft genome sequence of 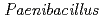 sp. strain TCA20 totals 5,631,463 bp in length and is composed of 33 large contigs (
sp. strain TCA20 totals 5,631,463 bp in length and is composed of 33 large contigs (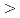 500 bp) that was deposited at DDBJ/EMBL/Genbank under the accession no. BBIW00000000. The annotation of the draft genome sequence shows that
500 bp) that was deposited at DDBJ/EMBL/Genbank under the accession no. BBIW00000000. The annotation of the draft genome sequence shows that 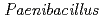 sp. strain TCA20 has a calcium-specific calcium/proton antiporter, ChaA, and a P-type calcium-transporting ATPase, YloB, was identified as calcium transporters. These transporters may also be important for the growth of
sp. strain TCA20 has a calcium-specific calcium/proton antiporter, ChaA, and a P-type calcium-transporting ATPase, YloB, was identified as calcium transporters. These transporters may also be important for the growth of 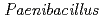 sp. strain TCA20 under the high concentration of calcium ions.
sp. strain TCA20 under the high concentration of calcium ions.
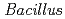 sp. strain TS-2, isolated from a jumping spider
sp. strain TS-2, isolated from a jumping spider藤浪 俊*; 武田 喜代子*; 小野寺 威文*; 佐藤 勝也; 佐野 元彦*; 鳴海 一成*; 伊藤 政博*
Genome Announcements (Internet), 2(3), p.e00458-14_1 - e00458-14_2, 2014/05
The potassium-dependent alkaliphilic 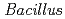 sp. strain TS-2 was isolated from the mashed extract of a jumping spider, and its draft genome sequence was obtained. Comparative genomic analysis with a previously sequenced sodium-dependent alkaliphilic
sp. strain TS-2 was isolated from the mashed extract of a jumping spider, and its draft genome sequence was obtained. Comparative genomic analysis with a previously sequenced sodium-dependent alkaliphilic 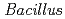 species may reveal potassium-dependent alkaline adaptation mechanisms. The draft genome sequence of
species may reveal potassium-dependent alkaline adaptation mechanisms. The draft genome sequence of 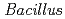 sp. strain TS-2 was 4,360,646 bp in total length, comprised 58 large contigs (
sp. strain TS-2 was 4,360,646 bp in total length, comprised 58 large contigs (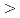 500 bp) that was deposited at DDBJ/EMBL/Genbank under the accession number BAWL00000000. In the draft genome sequence of
500 bp) that was deposited at DDBJ/EMBL/Genbank under the accession number BAWL00000000. In the draft genome sequence of 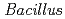 sp. strain TS-2, two sets of
sp. strain TS-2, two sets of 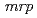 genes, which encode multisubunit secondary cation/proton antiporter-3 family proteins, were annotated. The Mrp complex acts as an Na
genes, which encode multisubunit secondary cation/proton antiporter-3 family proteins, were annotated. The Mrp complex acts as an Na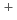 /H
/H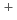 antiporter in typical alkaliphilic
antiporter in typical alkaliphilic 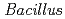 species and plays a critical role in the sodium-dependent alkaline adaptation mechanism.
species and plays a critical role in the sodium-dependent alkaline adaptation mechanism.